A 空间蛋白质组学理论与实践
马克斯普朗克生物化学研究所(Matthias Mann 团队)、西湖大学以及西湖欧米专业团队教学,从蛋白质组学基础入门,系统讲解DVP、FAXP等前沿空间蛋白技术原理与应用,涵盖组织膨胀、样本处理、质谱采集全流程。
B 蛋白质组学数据分析
聚焦质谱数据解析与AI建模,从原始数据质控、差异分析、功能注释到DIA-BERT搜库平台,覆盖多种主流分析策略与工具,打造从数据到科研发现的闭环能力体系。
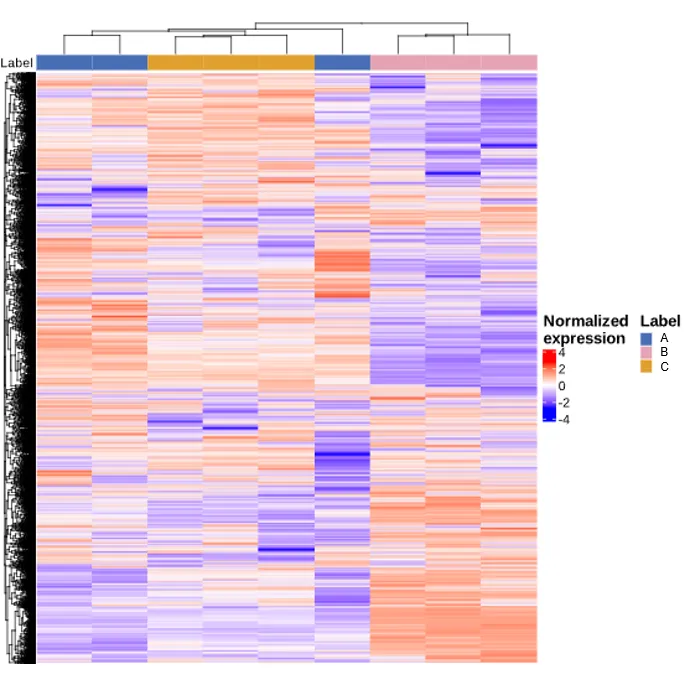




1. 蛋白质组质谱原始数据解析
本课程围绕蛋白质组质谱原始数据的解析,系统介绍了定量蛋白质组学的基本概念、主流技术及常用数据分析软件。
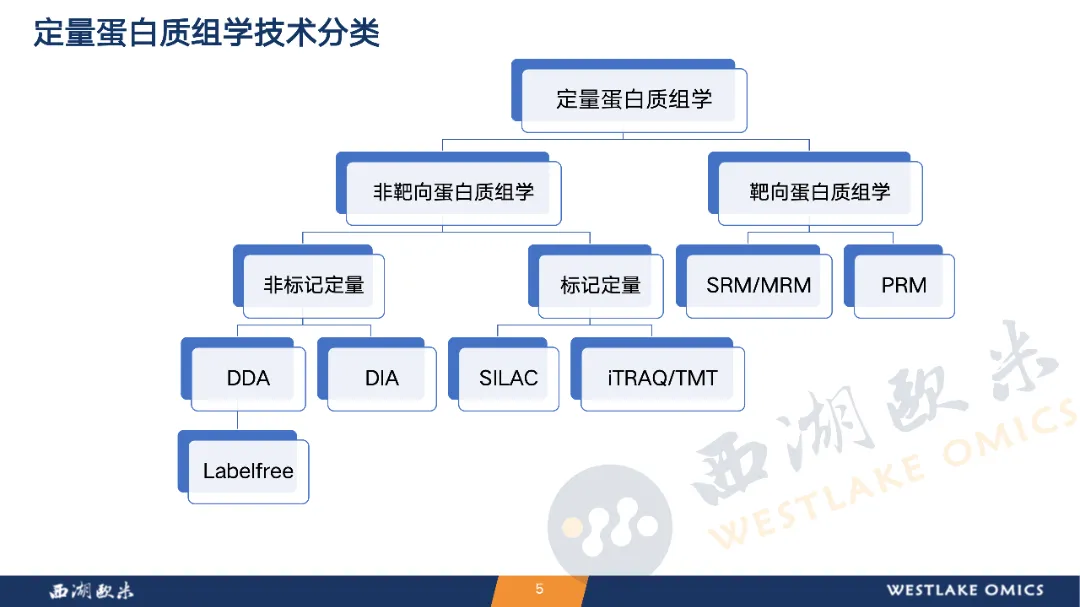
课程首先讲解了DDA(数据依赖采集)、DIA(数据非依赖采集)和靶向蛋白质组学等多种质谱技术的原理、优劣及适用场景;随后对iTRAQ/TMT标记定量与SILAC等方法进行了比较分析。
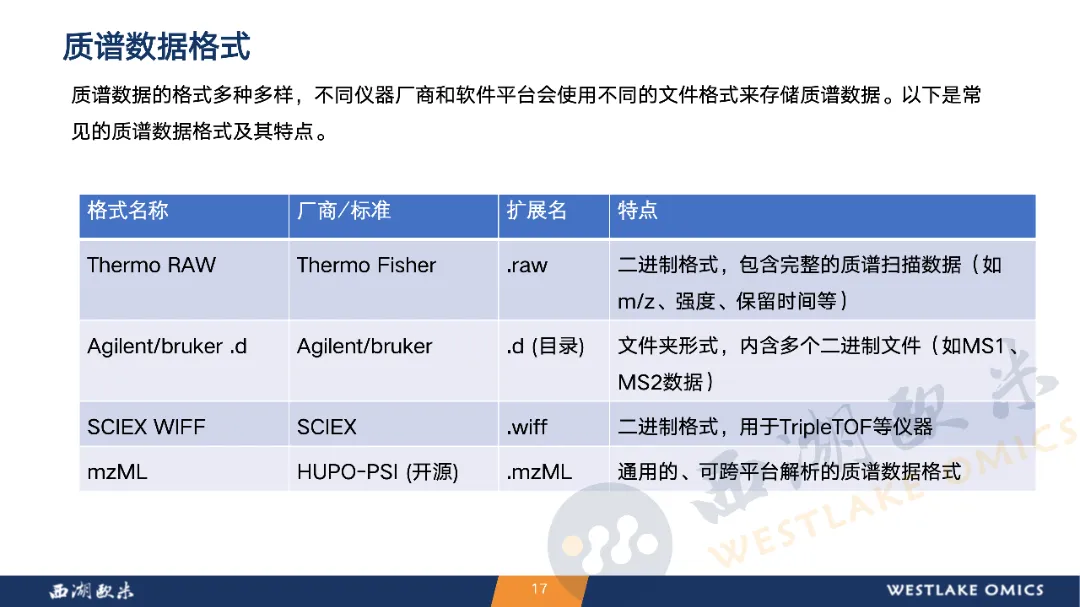
在软件工具方面,课程详细介绍了MaxQuant、FragPipe、DIA-NN、Spectronaut等用于不同类型数据的处理流程,并使用FragPipe、DIA-NN进行了操作演示。此外,还涉及质谱数据格式转换、蛋白数据库(如UniProt)使用及谱库构建方法。课程兼具理论阐释与实操指导,为学员全面理解和掌握质谱数据定量分析奠定了基础。

2. R语言基础与AI辅助编程
本课程围绕R语言在生物信息学中的基础应用及AI辅助编程展开,分为两个部分:
第一部分介绍了R语言与RStudio的基本概念、常见数据类型与数据结构,以及如何安装和使用CRAN、Bioconductor等资源包。课程还讲解了变量赋值、文件读写、常用函数、数据操作和可视化等核心技能,为学员打下扎实的编程基础。
第二部分聚焦AI工具(如Copilot、ChatGPT)在R编程中的应用,涵盖代码补全、语法解释及实际编程示例,帮助学员提升编程效率和理解能力。课程结合实例演示与练习,增强实战能力,适合初学者快速入门R语言并掌握AI辅助开发技巧。



3. 蛋白质组数据预处理
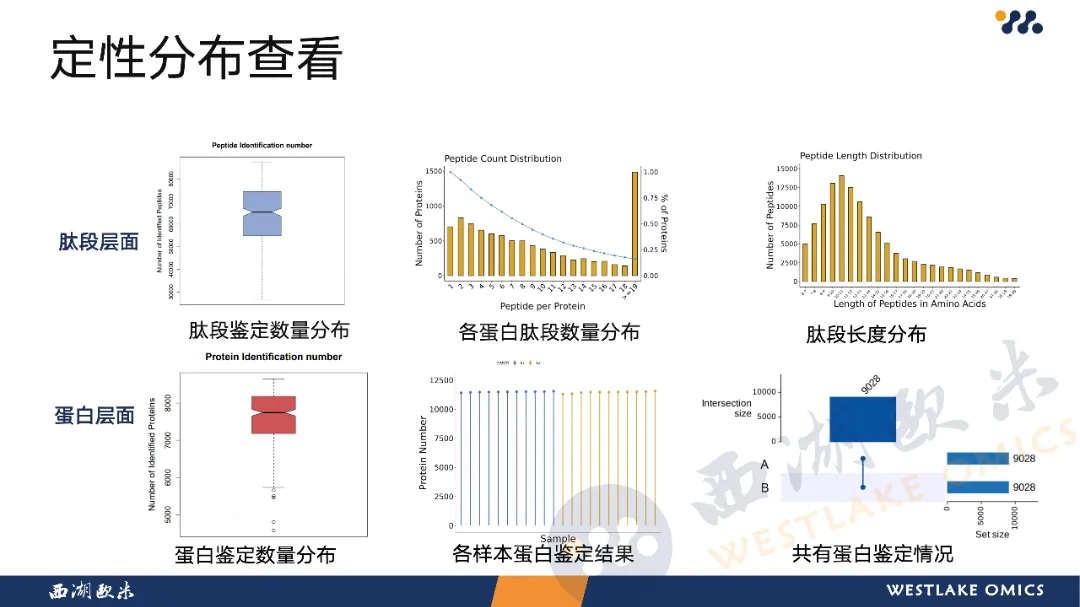
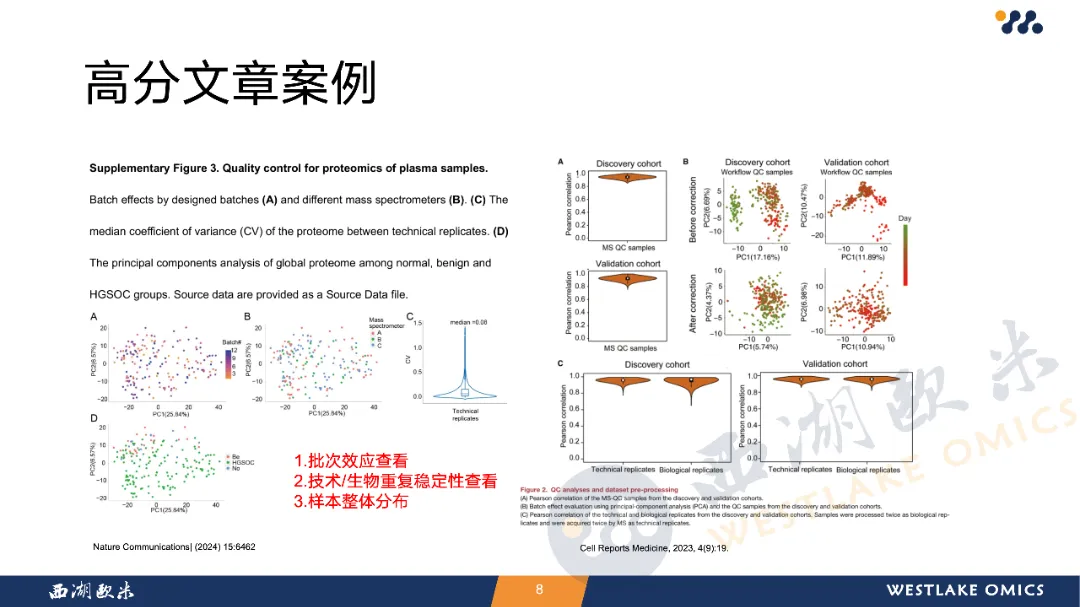
本课程聚焦于蛋白质组学数据的预处理,旨在提升数据质量和分析可靠性。
课程首先介绍了数据质控的重要性,然后介绍了从不同角度评估质控好坏的方法,包括定性定量指标的可视化、QC样本的稳定性评估、异常样本和批次效应检测。随后深入讲解了预处理流程,如异常样本和高缺失率蛋白的筛除、缺失值填充(依据缺失机制选择合适方法,如KNN、SVD、最小值填充等)、数据归一化(log2转换、Quantile、Z-score等)以及批次效应校正(如ComBat、removeBatchEffect)。
通过引用高水平文献案例,课程结合实际数据分析策略,强调预处理在保障蛋白质组学研究有效性中的关键作用,适用于科研人员掌握标准化分析流程。

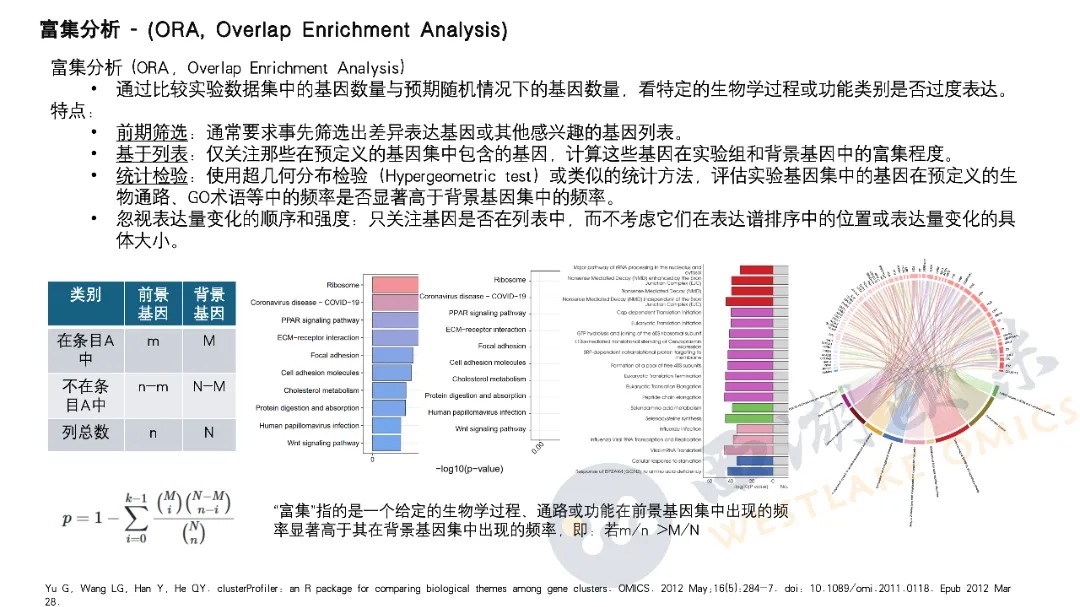
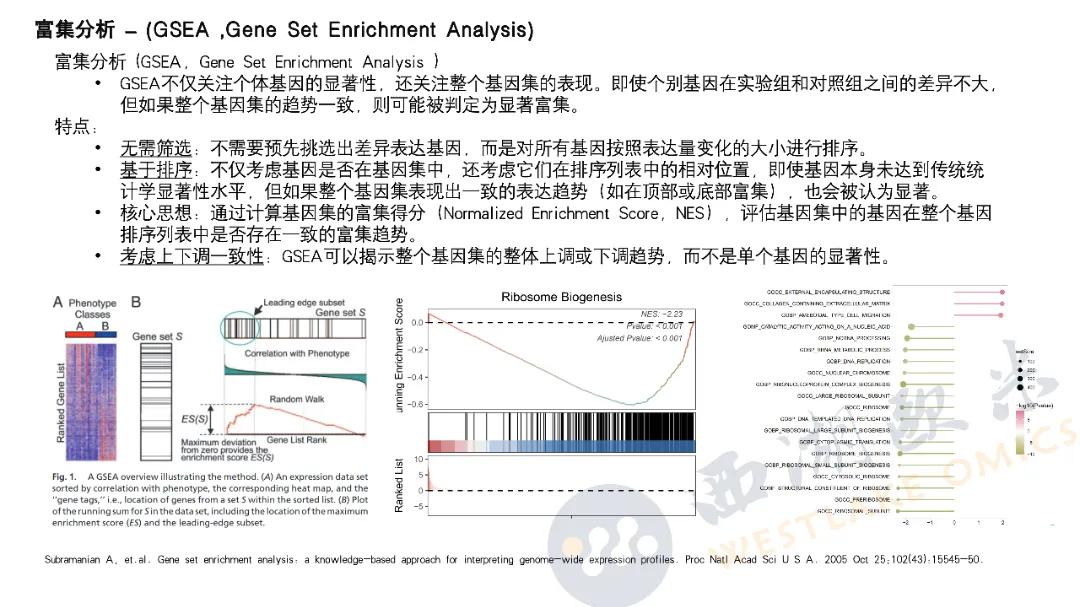
4. 富集分析&蛋白互作网络分析
本课程系统讲解了富集分析与蛋白互作网络分析的原理、方法及工具。
首先介绍了富集分析的两大主流方法:ORA(基于基因列表的过度表达分析)和GSEA(基于全排序基因集趋势分析),并比较了它们的特点与适用场景。课程进一步介绍了多种富集分析工具,如Metascape、g\:Profiler、Enrichr-KG、WebGestalt 和 Pathview,展示了其在功能注释、通路分析及可视化方面的优势。
随后,课程讲解了蛋白互作网络(PPI)分析的意义与方法,重点介绍了STRING、GeneMANIA 和 STITCH 等常用数据库与平台,帮助学员构建和解析蛋白-蛋白及蛋白-化合物互作图谱。课程内容结合分析实操,增强学员对分析方法的理解及数据挖掘能力。
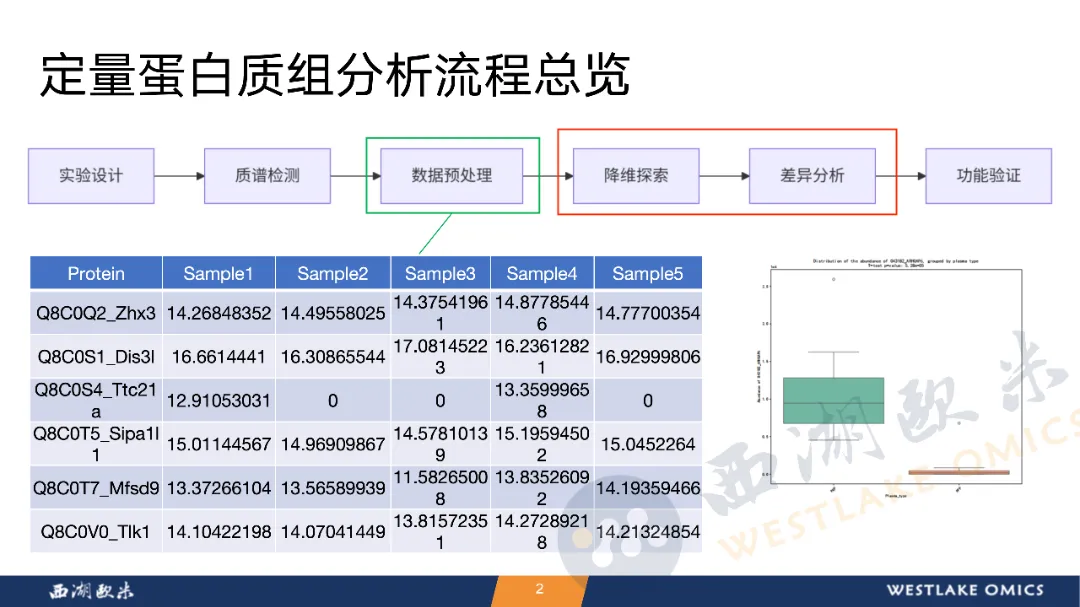
5. 蛋白组学数据差异分析与数据降维
本课程聚焦蛋白质组学中的差异分析与数据降维,帮助学员掌握高维数据的可视化与关键特征识别。
首先介绍了PCA、t-SNE和UMAP等常见降维方法的原理、优劣和适用场景,并强调降维前需标准化与填补缺失值。随后讲解差异分析的核心流程,包括差异倍数(FoldChange)与显著性检验(t检验、ANOVA),以及多重比较校正方法(如BH、Bonferroni)。课程还涵盖多组比较的Post Hoc分析策略(如Tukey、Games-Howell等)与聚类方法(如mfuzz)用于多组比较时的表达趋势归类。
最后通过火山图、热图、Upset图等多种可视化手段,展示差异分析结果,为蛋白质组数据解读提供全面工具。


在培训的尾声,来自西湖大学医学院蛋白质组复杂科学实验室和西湖欧米AI团队的讲师还介绍了Transformer模型的原理及其在蛋白质组学中的实际应用。
本课程通俗讲解了Transformer模型的原理和结构,并对比了BERT与GPT两种模型的差异,展示了它在自然语言处理和生物医学等领域的应用,帮助学员建立从理论到实战的整体认知。
课程随后介绍了DIA-BERT,它在Transformer的基础上对大规模的DIA质谱数据进行预训练,提升了蛋白质识别的深度和准确性,特别是在低丰度蛋白识别方面表现出色。
欢迎您以任何方式宣传我们的蛋白组学培训课程。

A 空间蛋白质组学理论与实践
马克斯普朗克生物化学研究所(Matthias Mann 团队)、西湖大学以及西湖欧米专业团队教学,从蛋白质组学基础入门,系统讲解DVP、FAXP等前沿空间蛋白技术原理与应用,涵盖组织膨胀、样本处理、质谱采集全流程。
B 蛋白质组学数据分析
聚焦质谱数据解析与AI建模,从原始数据质控、差异分析、功能注释到DIA-BERT搜库平台,覆盖多种主流分析策略与工具,打造从数据到科研发现的闭环能力体系。





1. 蛋白质组质谱原始数据解析
本课程围绕蛋白质组质谱原始数据的解析,系统介绍了定量蛋白质组学的基本概念、主流技术及常用数据分析软件。
课程首先讲解了DDA(数据依赖采集)、DIA(数据非依赖采集)和靶向蛋白质组学等多种质谱技术的原理、优劣及适用场景;随后对iTRAQ/TMT标记定量与SILAC等方法进行了比较分析。
在软件工具方面,课程详细介绍了MaxQuant、FragPipe、DIA-NN、Spectronaut等用于不同类型数据的处理流程,并使用FragPipe、DIA-NN进行了操作演示。此外,还涉及质谱数据格式转换、蛋白数据库(如UniProt)使用及谱库构建方法。课程兼具理论阐释与实操指导,为学员全面理解和掌握质谱数据定量分析奠定了基础。



2. R语言基础与AI辅助编程
本课程围绕R语言在生物信息学中的基础应用及AI辅助编程展开,分为两个部分:
第一部分介绍了R语言与RStudio的基本概念、常见数据类型与数据结构,以及如何安装和使用CRAN、Bioconductor等资源包。课程还讲解了变量赋值、文件读写、常用函数、数据操作和可视化等核心技能,为学员打下扎实的编程基础。
第二部分聚焦AI工具(如Copilot、ChatGPT)在R编程中的应用,涵盖代码补全、语法解释及实际编程示例,帮助学员提升编程效率和理解能力。课程结合实例演示与练习,增强实战能力,适合初学者快速入门R语言并掌握AI辅助开发技巧。



3. 蛋白质组数据预处理
本课程聚焦于蛋白质组学数据的预处理,旨在提升数据质量和分析可靠性。
课程首先介绍了数据质控的重要性,然后介绍了从不同角度评估质控好坏的方法,包括定性定量指标的可视化、QC样本的稳定性评估、异常样本和批次效应检测。随后深入讲解了预处理流程,如异常样本和高缺失率蛋白的筛除、缺失值填充(依据缺失机制选择合适方法,如KNN、SVD、最小值填充等)、数据归一化(log2转换、Quantile、Z-score等)以及批次效应校正(如ComBat、removeBatchEffect)。
通过引用高水平文献案例,课程结合实际数据分析策略,强调预处理在保障蛋白质组学研究有效性中的关键作用,适用于科研人员掌握标准化分析流程。



4. 富集分析&蛋白互作网络分析
本课程系统讲解了富集分析与蛋白互作网络分析的原理、方法及工具。
首先介绍了富集分析的两大主流方法:ORA(基于基因列表的过度表达分析)和GSEA(基于全排序基因集趋势分析),并比较了它们的特点与适用场景。课程进一步介绍了多种富集分析工具,如Metascape、g\:Profiler、Enrichr-KG、WebGestalt 和 Pathview,展示了其在功能注释、通路分析及可视化方面的优势。
随后,课程讲解了蛋白互作网络(PPI)分析的意义与方法,重点介绍了STRING、GeneMANIA 和 STITCH 等常用数据库与平台,帮助学员构建和解析蛋白-蛋白及蛋白-化合物互作图谱。课程内容结合分析实操,增强学员对分析方法的理解及数据挖掘能力。



5. 蛋白组学数据差异分析与数据降维
本课程聚焦蛋白质组学中的差异分析与数据降维,帮助学员掌握高维数据的可视化与关键特征识别。
首先介绍了PCA、t-SNE和UMAP等常见降维方法的原理、优劣和适用场景,并强调降维前需标准化与填补缺失值。随后讲解差异分析的核心流程,包括差异倍数(FoldChange)与显著性检验(t检验、ANOVA),以及多重比较校正方法(如BH、Bonferroni)。课程还涵盖多组比较的Post Hoc分析策略(如Tukey、Games-Howell等)与聚类方法(如mfuzz)用于多组比较时的表达趋势归类。
最后通过火山图、热图、Upset图等多种可视化手段,展示差异分析结果,为蛋白质组数据解读提供全面工具。



在培训的尾声,来自西湖大学医学院蛋白质组复杂科学实验室和西湖欧米AI团队的讲师还介绍了Transformer模型的原理及其在蛋白质组学中的实际应用。
本课程通俗讲解了Transformer模型的原理和结构,并对比了BERT与GPT两种模型的差异,展示了它在自然语言处理和生物医学等领域的应用,帮助学员建立从理论到实战的整体认知。
课程随后介绍了DIA-BERT,它在Transformer的基础上对大规模的DIA质谱数据进行预训练,提升了蛋白质识别的深度和准确性,特别是在低丰度蛋白识别方面表现出色。
欢迎您以任何方式宣传我们的蛋白组学培训课程。








