4月22日,威斯康星大学麦迪逊分校和霍华德·休斯医学研究所团队的研究人员在 Nature Methods 发表了一项关于空间组学技术发展的研究研究,介绍了一种新型的组织膨胀质谱成像方法(TEMI,tissue-expansion method compatible with MALDI-MS imaging)。

图1 文章截图
提纲挈领
研究开发了一种兼容质谱成像的组织膨胀技术(TEMI),在不破坏组织内生物分子的前提下实现了单细胞尺度的高分辨率、多分子类型空间成像,显著提升了MALDI质谱成像的空间解析能力和适用范围。
TEMI可在单细胞尺度下同时成像脂质、代谢物、蛋白质和N-聚糖,具备高通量和高保真度的特点。
通过在小鼠小脑、肿瘤及多种器官中的应用,研究人员揭示了组织内部的分子异质性,验证了TEMI在空间多组学研究中的广泛适用性和精度优势,为理解组织结构与功能之间的关系提供了新平台。
在多细胞生物体中,生物分子(如脂质、代谢物、蛋白质和糖类)的空间分布对于理解其生理功能和病理过程至关重要。尽管空间转录组技术在核酸成像方面取得了巨大进展,但目前在非标记条件下对其他分子类型进行高分辨率、大通量空间成像仍面临挑战。传统的质谱成像(MSI)因分辨率受限等问题,在实际生物医学研究中应用受到制约。
为克服上述限制,研究团队开发了TEMI,该方法通过在不破坏生物分子的前提下对组织进行物理膨胀,从而显著提升质谱成像的空间分辨率,实现对多种分子的原位高通量分析。
与现有方法不同,TEMI避免了蛋白质变性等剧烈处理,保留了组织中生物分子的天然状态,提高了成像灵敏度和分子定位的准确性。
研究团队进一步表明,该方法可用于脂质组学、代谢组学、蛋白质组学及N-聚糖组学的空间成像分析,在小鼠组织、肿瘤样本等多种生物医学模型中展现出强大的分辨能力和普适性。
01
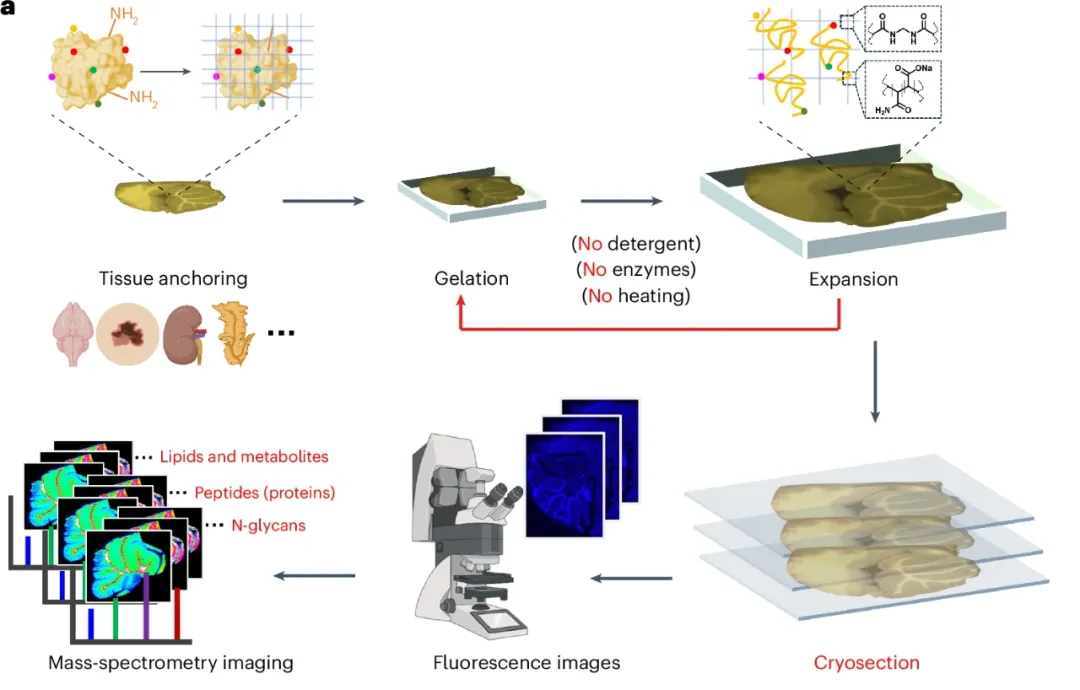
TEMI组织膨胀质谱成像方法的建立与优化
研究团队首先针对传统组织膨胀方法在MSI应用中的不足提出了改进策略。常规膨胀方法依赖高温、去污剂或蛋白酶处理以实现组织水凝胶的均匀膨胀,但这类剧烈的处理容易导致信号丢失和分子空间错位。
研究人员假设,在不进行蛋白质变性的前提下(denaturation-free expansion),利用组织中天然态蛋白作为锚定基质,可更有效保留脂质、代谢物、肽段和糖类等分子。通过对冷冻切片条件的优化(如30%蔗糖浓度与适当厚度选择)以及高强度水凝胶的使用,研究者成功构建出可被MALDI-MS兼容分析的组织样本。
此外,通过比较不同处理方法下脂质的提取与保留情况,结果表明无蛋白酶处理的组织保留效果远优于传统方法。后续的膨胀实验进一步验证,在无变性处理条件下,即使仅1.5倍膨胀,也能显著提升图像结构解析度。
在经过两轮嵌胶后,组织可稳定膨胀至2.5倍,并在MSI成像中表现出与未膨胀样本一致的质量图谱,未见明显化学扩散或污染背景。
02
TEMI揭示小脑分层结构中的脂质分布特异性
研究利用TEMI方法对小鼠小脑组织进行约2.5倍和3.5倍膨胀后,成功以高空间分辨率成像揭示了不同小脑层(分子层、颗粒层和白质层)中脂质分布的特异性。
不同区域富集有特定的磷脂分子,反映出组织结构与分子构成的紧密关联。进一步提高膨胀倍数后,成像分辨率达~2.9 µm,首次在MSI图像中清晰识别出浦肯野细胞(purkinje cells)及其特异性脂质指纹,表明TEMI显著提升了脂质空间异质性的可视化能力,优于传统质谱成像方法。
03
小脑中代谢物的空间分布图谱分析
研究团队进一步将TEMI拓展至小脑组织中的代谢物成像分析。通过在2.5倍膨胀组织中应用特定MALDI基质,研究者在正负离子模式下共检测到187种代谢特征。
多个代谢物在小脑的不同功能层之间展现出显著的空间分布差异,而这些差异在未膨胀组织中则难以识别。部分代谢物如胆碱、己糖磷酸、OEA和2-AG已被明确注释,但由于当前缺乏与MALDI-MSI相配套的完整代谢物参考库,其余多数仍未能准确标注。
这一结果不仅验证了小脑在代谢层面存在显著异质性,也突显TEMI在代谢组空间解析中的应用价值。
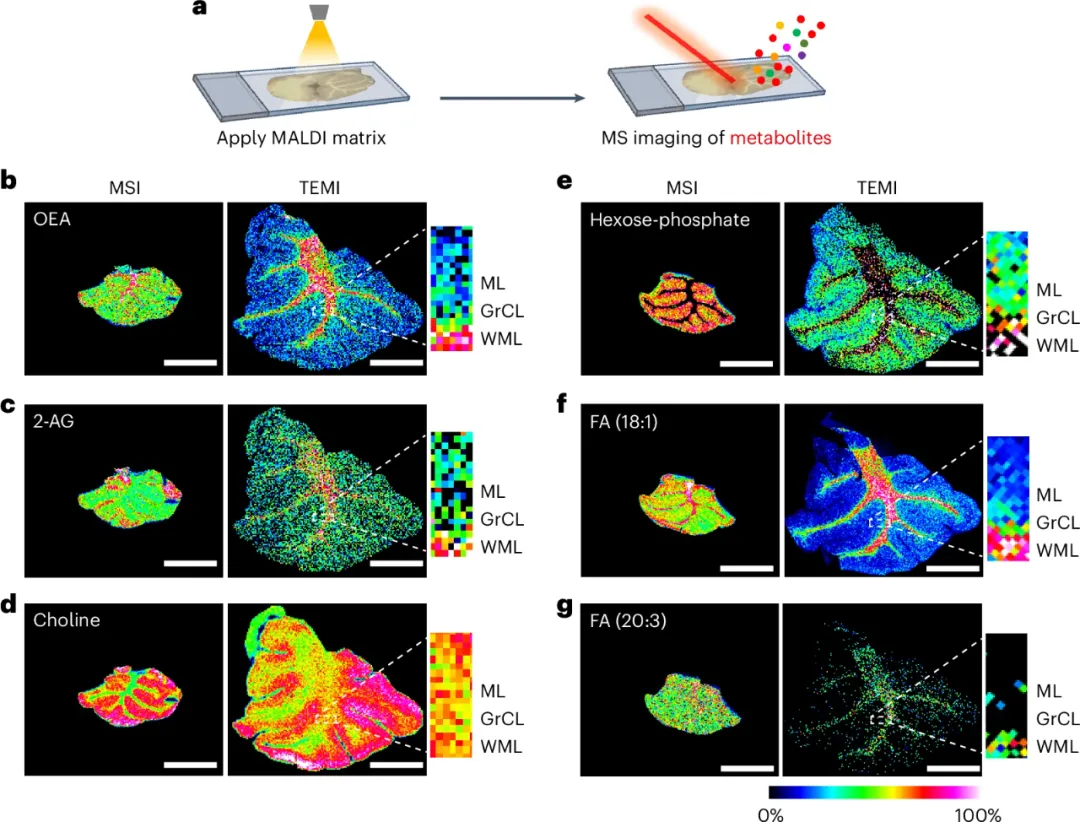
图4 TEMI分析小脑代谢物分布
04
利用TEMI绘制小脑蛋白质和N-聚糖图谱
研究者进一步膨胀TEMI在蛋白质组与糖组的应用,利用约3.5倍膨胀的小脑切片,通过原位胰蛋白酶消化结合MSI与高分辨串联质谱,成功鉴定出包括MBP(Myelin Basic Protein,髓鞘碱性蛋白)与组蛋白H2B在内的57种肽段。
这些蛋白质在组织中的分布具有明显的区域特异性,例如MBP富集于白质层,H2B富集于颗粒层。通过PC-MT标记抗体的IHC-MALDI-MSI分析,也进一步确认了上述蛋白分布的空间特征。
与此同时,研究者检测出多达29种N-聚糖,其中部分表现出中度的组织区分性,如HexNAc2Hex5。以上结果表明TEMI不仅适用于小分子与脂质,也能支持复杂大分子的空间成像分析。
05
利用TEMI揭示肿瘤的高度代谢异质性
研究人员将TEMI方法应用于小鼠黑色素瘤模型,经过三轮组织膨胀后实现约3.5倍膨胀,并使用50 µm激光扫描进行质谱成像。
结果显示,膨胀组织在空间分辨率上明显优于未膨胀组织,能够清晰区分代谢特征如PC (36:1) 和 PC (38:4) 的空间分布。通过质谱谱图聚类分析,TEMI可将肿瘤组织细分为多达21个代谢区域,而未膨胀组织仅能分出3个区域,揭示了肿瘤组织高度的代谢异质性,证明TEMI在癌症研究中的潜在应用价值。
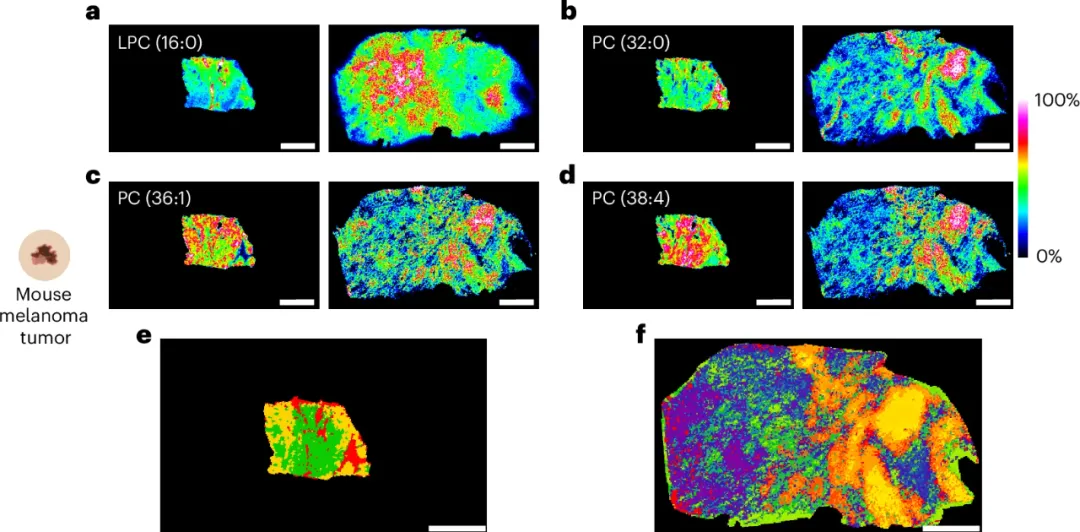
图5 利用TEMI揭示小鼠肿瘤的代谢异质性
06
TEMI在多器官分子成像中的广泛适用性
除小脑与肿瘤外,研究还将TEMI应用于小鼠肾脏和胰腺等器官,实现了脂质分子的高分辨空间定位,展示出该技术在多种组织类型中的普适性。各器官在膨胀过程中所产生的形变误差均小于12%,表明在保持组织结构真实性方面具备良好的稳定性。这一结果验证了TEMI不仅适用于脑组织或肿瘤模型,还可广泛用于多器官、多分子类型的空间多组学研究,具有极高的应用拓展潜力。
综上所述,TEMI提供了一种成本较低、适配性强且生物分子保护能力优越的质谱成像新平台,显著推动了空间多组学的研究进程。
未来,随着锚定策略与多重免疫标记技术的进一步优化,TEMI有望在基础生物学、疾病机制研究及临床诊断中发挥更广泛的应用价值。
论文链接:
https://www.nature.com/articles/s41592-025-02664-9