
2023年11月30日,西湖大学蛋白质组大数据实验室郭天南团队在bioRxiv上发表预印本文章,题为metaExpertPro: a computational workflow for metaproteomics spectral library construction and data-independent acquisition mass spectrometry data analysis。
预印本文章介绍了一个名为metaExpertPro的计算工作流程,为宏蛋白质组学谱库构建、肽段和蛋白的鉴定和定量、功能和分类群注释提供了一站式分析平台,尤其为基于数据非依赖性质谱采集(DIA-MS)的宏蛋白质学数据分析提供了准确、高效的解决方案。
过去几年中,由于微生物在人类健康中(尤其是在营养、新陈代谢和免疫方面)的关键作用,微生物群落和功能引起了越来越多的关注。宏基因组学和代谢组学已广泛应用于肠道微生物研究,但它们在解析微生物组的蛋白功能方面存在一些局限。
宏蛋白组学(Metaproteomics)是一个新兴的研究领域,在量化整个微生物群落中真正表达的蛋白质方面具有独特的优势。然而宏蛋白质组学数据分析,尤其是对于大规模的DIA-MS数据分析,仍是一项挑战。
传统的质谱工具如 X!Tandem、OMSSA 等主要适用于数据依赖性质谱采集(DDA-MS)数据,对于大规模 metaproteomics 数据的计算效率不高。为了提高计算效率,专门用于宏蛋白组分析的软件如 metaLab、MetaProteomeAnalyzer(MPA)等已经被开发,但它们也仅适用于 DDA-MS 数据。作为高重复性、高通量、高深度的蛋白组学方法,DIA-MS 需要更复杂的数据分析。
因此,研究团队开发了新的宏蛋白组数据分析工作流metaExpertPro,可高效构建谱库、分析DIA-MS数据,并进行分类和功能数据的注释。
具体来讲,metaExpertPro工作流程包括四个阶段:基于 DDA-MS 的谱库生成、基于 DIA-MS 的多肽和蛋白的定量、功能和分类群注释以及定量矩阵生成,它同时兼容 Thermo Fisher Orbitrap 或 Bruker 质谱仪的 DDA-MS 和 DIA-MS 数据。
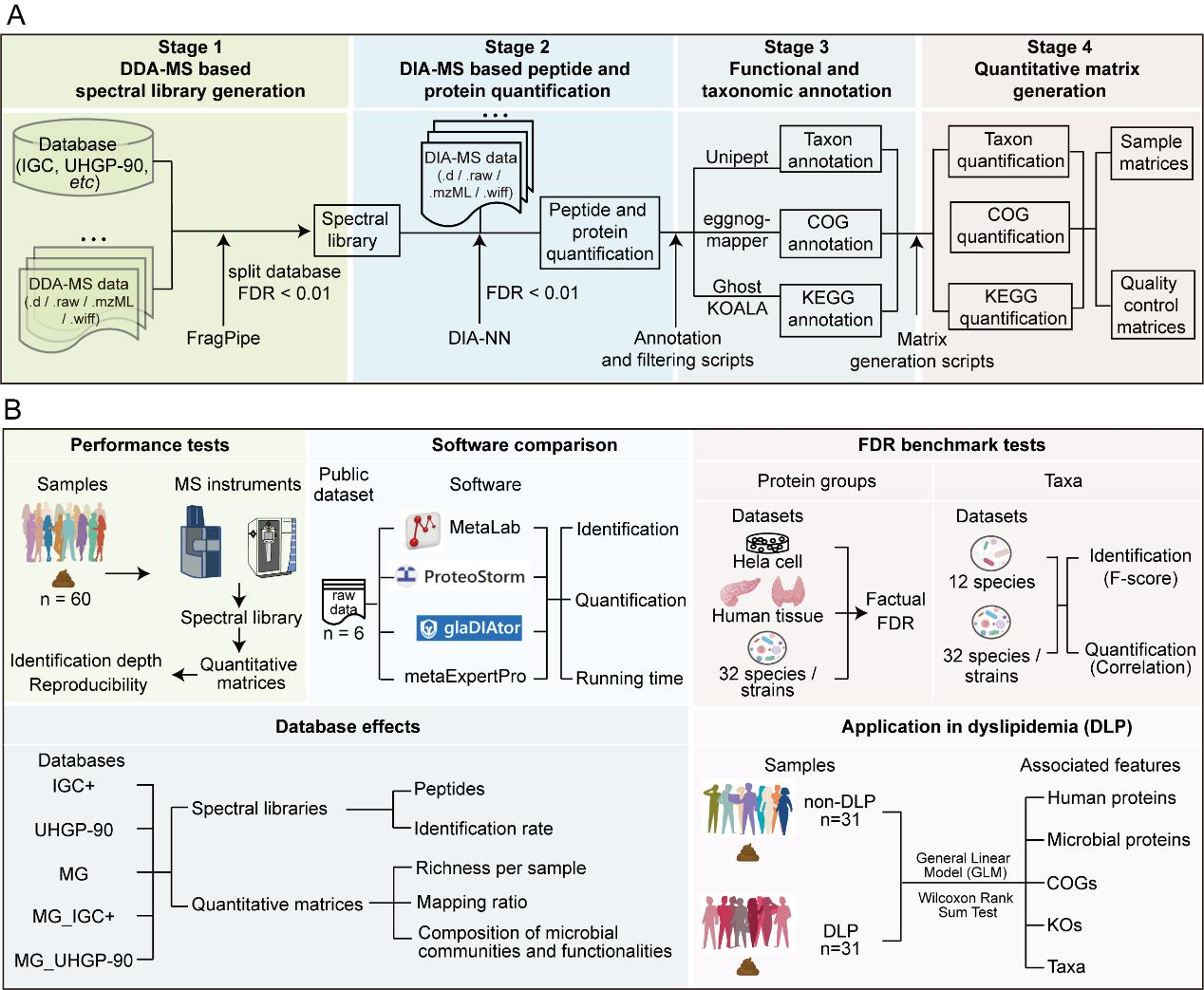
图1 metaExpertPro 的计算工作流程和性能测试概览
metaExpertPro工作流程详解
第一阶段 使用 FragPipe 软件生成谱库
● 利用FragPipe进行DDA-MS谱库生成,采用MSFragger的数据库拆分参数,将原始数据库(如人类肠道微生物基因目录数据库(IGC)和人类胃肠蛋白质数据库(UHGP))拆分成多个数据库,以减小计算内存需求。
● 对每个拆分的数据库进行DDA-MS原始数据搜索,生成pepXML和pin文件。
● 所有DDA-MS原始数据的pepXML和pin文件进行合并,使用PeptideProphe进行PSM验证(保持相对低的factual FDR)。
第二阶段 DIA-NN用于肽段和蛋白质定量
● 使用DIA-NN软件进行DIA-MS数据文件中的肽段和蛋白质的鉴定和定量。
第三阶段 分类群注释和功能注释
● 分别使用 Unipept、eggnog-mapper 和 GhostKOALA 对分类群、COG 和 KEGG 进行注释,然后通过内部过滤脚本对注释结果进行过滤。
第四阶段 生成定量矩阵
● 最后生成包括人类肽、微生物肽、人类蛋白、微生物蛋白、COG、KO、COG类别、KO类别和分类群在内的九个层次的定量矩阵。
在人类粪便样本上进行的性能测试中,metaExpertPro在60分钟的单针diaPASEF采集中平均每个样本鉴定到45,000个肽段,表现出较高的鉴定深度、可重复性和一致性,相较于其他工具表现更出色(MetaLab、ProteoStorm和glaDIAtor)。在四项基准测试中,metaExpertPro保持了低于5%的蛋白质组水平的错误发现率(FDR)。
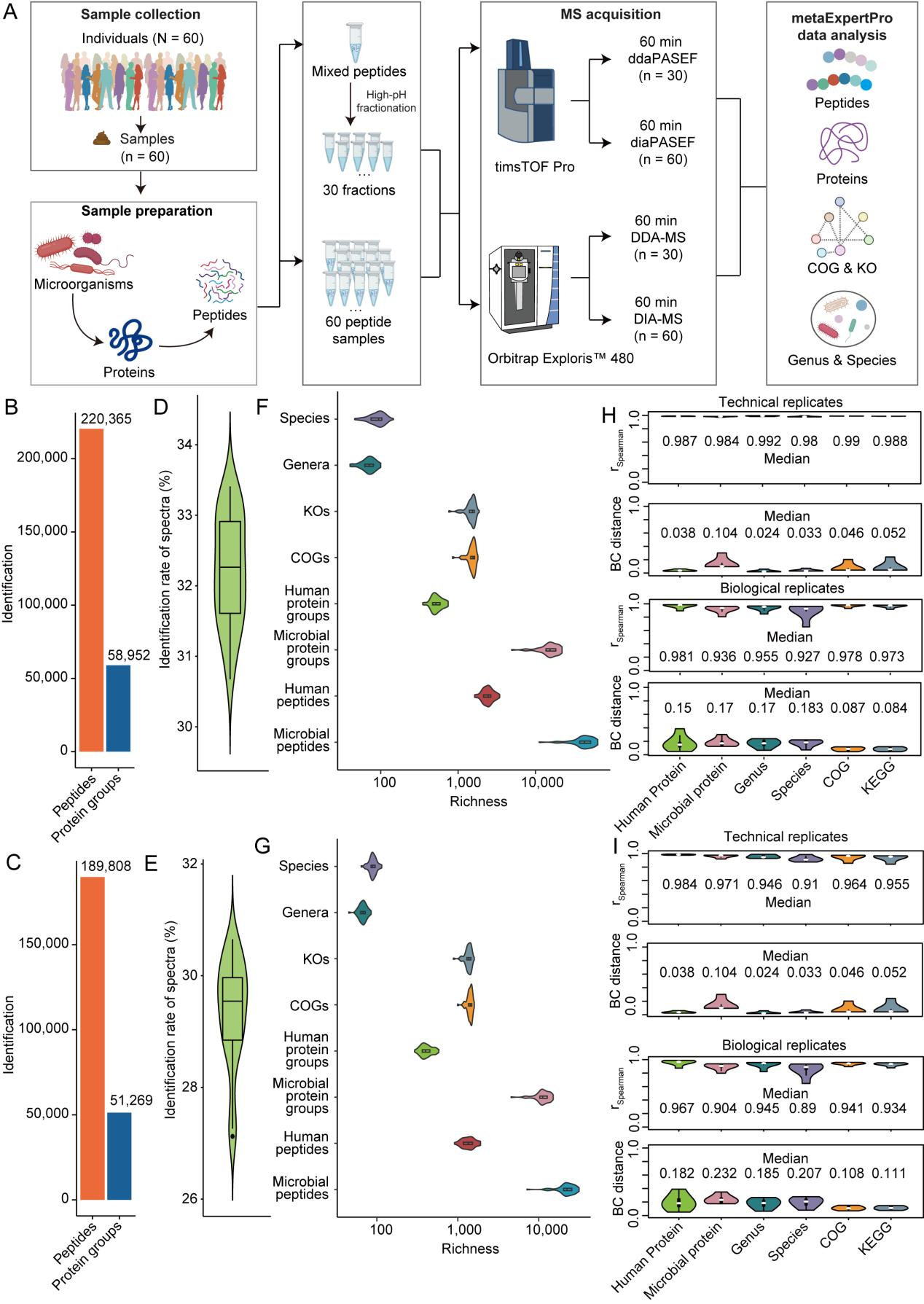
图2 metaExpertPro在人类粪便样本宏蛋白质组学分析中的深度鉴定和高度重现性
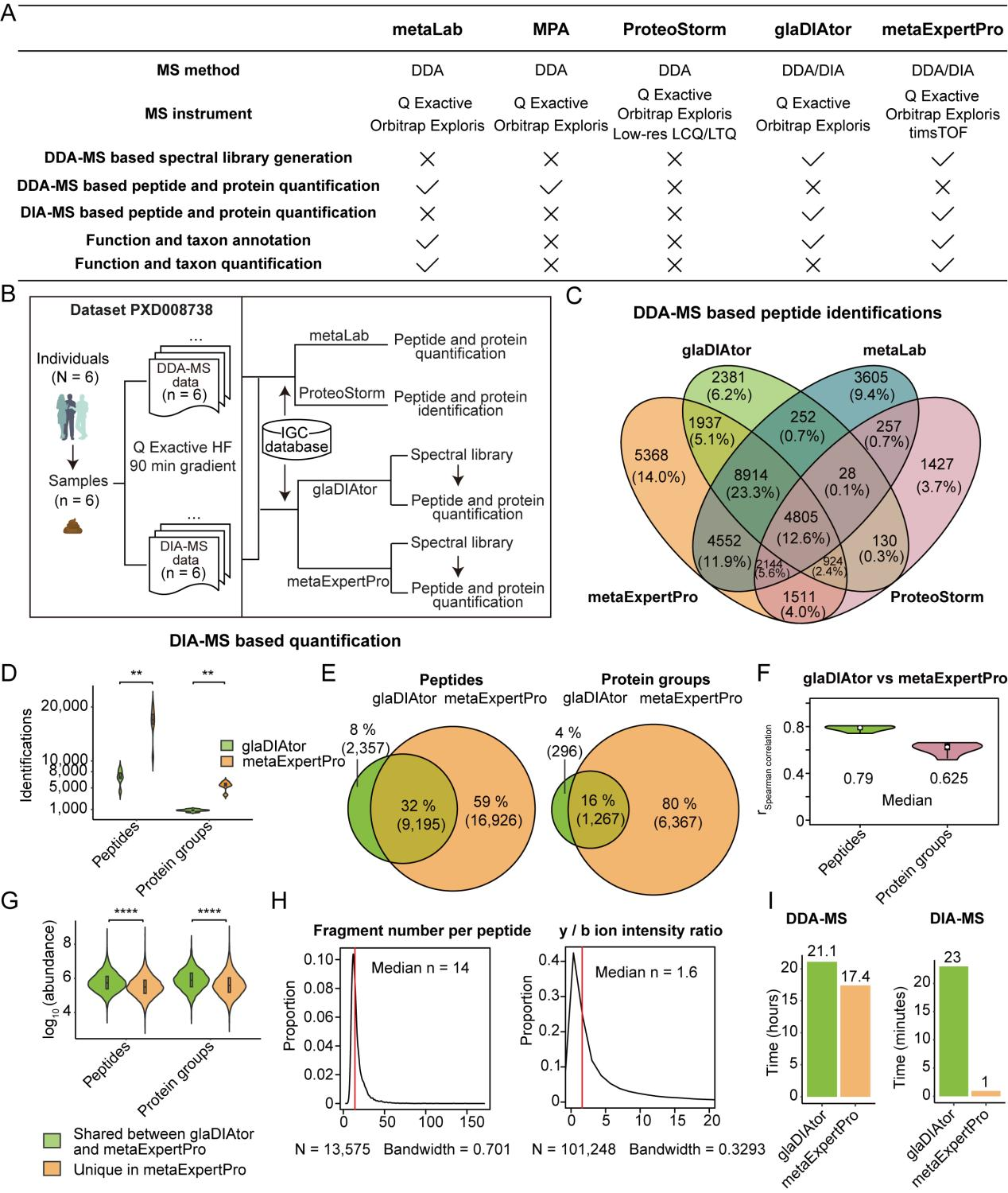
图3 metaExpertPro 与其他宏蛋白质组学软件工具的比较
此外,在基于公共人类肠道微生物蛋白质数据库(IGC和UHGP)的分析中,metaExpertPro展现了在蛋白质、分类群和功能水平的高度可重复性。在对高脂血症患者进行的宏蛋白质组学分析中,metaExpertPro揭示了微生物功能的特征性变化以及微生物与宿主之间的潜在相互作用。
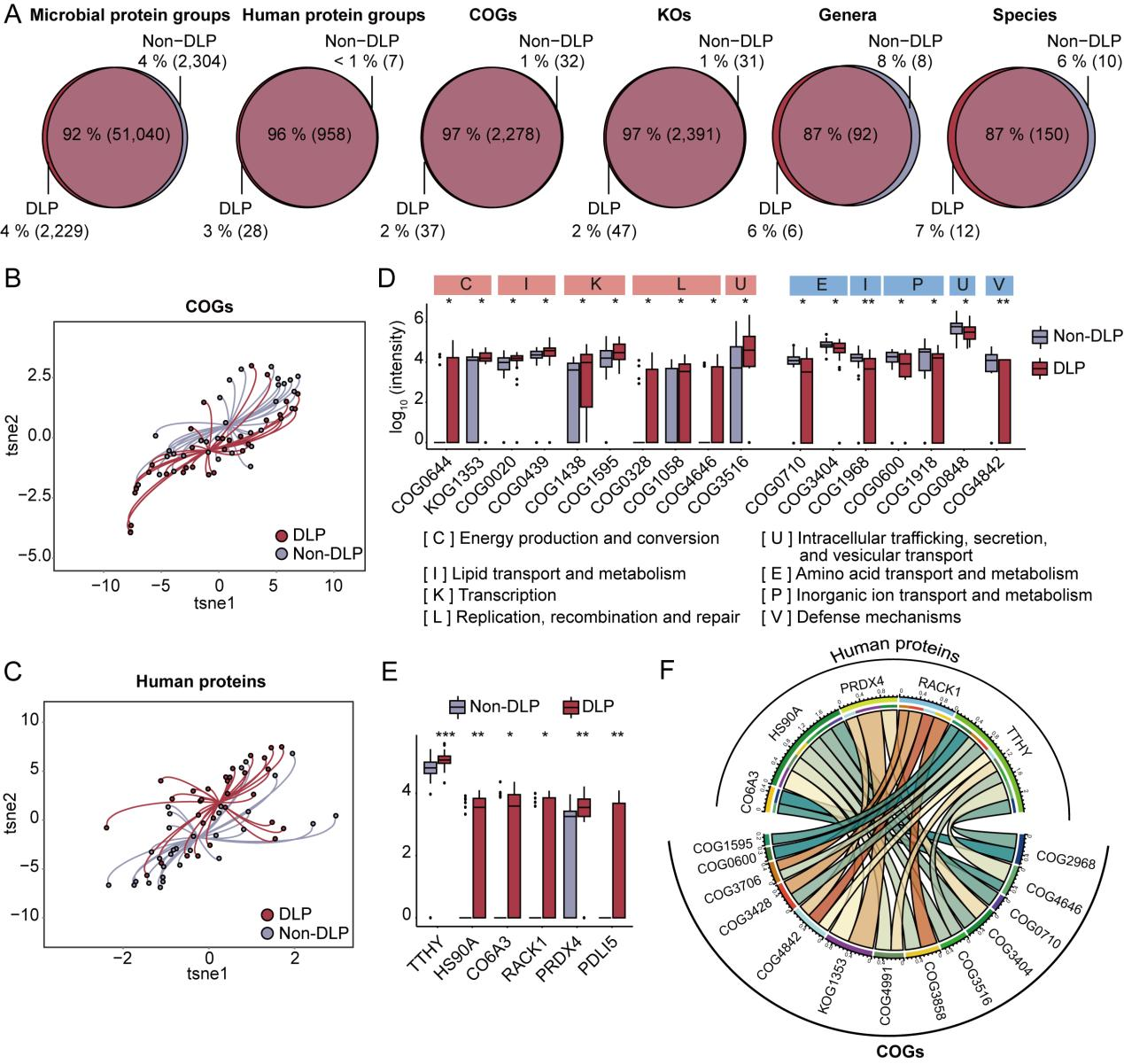
图4 基于 metaExpertPro 工作流程的与高血脂症(DLP)相关的蛋白质、功能和分类群
研究指出,由于样本的复杂性,宏蛋白组的数据分析存在一些固有的局限性,包括对数据库的高依赖性、低效的肽鉴定率、相对较低的分类鉴定分辨率和大量的计算机内存消耗。此外,对FDR的控制仍然是一个挑战,需要更有效的算法来区分高度相似的谱图,并采用更严格的FDR过滤方法来确保更准确的鉴定。
虽然metaExpertPro在两个公共肠道微生物蛋白质数据库上的结果表明,数据库对peptide、功能和分类的测量影响不大,但作者也指出,对其他基因目录数据库或其他类型的宏蛋白质组样本的结果可能会有所不同。
总体而言,metaExpertPro工作流程整合了高性能的蛋白质分析工具,为宏蛋白组数据分析提供了一种全面、高效的解决方案。其在宏蛋白质组学领域的性能和适用性方面取得了显著的进展,为深入了解微生物与宿主相互作用提供了有力工具。
论文链接:
https://www.biorxiv.org/content/10.1101/2023.11.29.569331v1








